- Published on
An AI system to help scientists write expert-level empirical software
- Authors

- Name
- Ryan Chung
- Name
- Google Gemini
E. Aygün et al., “An AI system to help scientists write expert-level empirical software,” Sep. 08, 2025, arXiv:2509.06503. https://arxiv.org/abs/2509.06503
Github: https://github.com/google-research/score
前言
想像一下,如果科學家不再需要花費數年時間去寫複雜的實驗程式,而是由 AI 在數小時或數天內自動生成並優化,甚至其表現還超越了人類專家。 這聽起來像是科幻小說,但這正是 Google 最新研究的核心突破。科學發現的循環往往被實驗所需軟體的「手動建立時間」所限制。 這項研究推出了一個結合了 大型語言模型(LLM) 與 樹狀搜尋(Tree Search, TS) 的 AI 系統, 專門用於系統性地創建「經驗軟體」(empirical software), 目標是最大化程式碼解決特定科學問題時所達到的「品質分數」(quality metric)。 接下來,我們將深入探討這個系統如何橫掃生物資訊學、流行病學、地理空間分析等多個領域的挑戰,並實現前所未有的科學加速。
科學的「軟體瓶頸」
幾乎每一個科學子領域、應用數學與工程現在都高度依賴軟體來推進研究。 我們將這種用於最大化可定義或可測量品質分數的軟體,稱為 經驗軟體 (Empirical Software)。 無論是預測天氣的模型參數化,還是分子動力學模擬,都屬於這類軟體。 然而,創建這些領域專屬的經驗軟體是極其耗時且困難的。 當科學家試圖用軟體來測試複雜假設時,他們很少能系統性地搜尋所有替代方法。 設計決策往往是基於直覺或便利性,而非窮盡式的實驗。 這種瓶頸嚴重限制了科學家能夠在合理時間內探索的可能性。 這促使研究人員思考:能否開發一個工具,能夠自動化且智慧地探索龐大的解決方案空間,從而打破這一限制?
AI 與樹狀搜尋的智慧結合
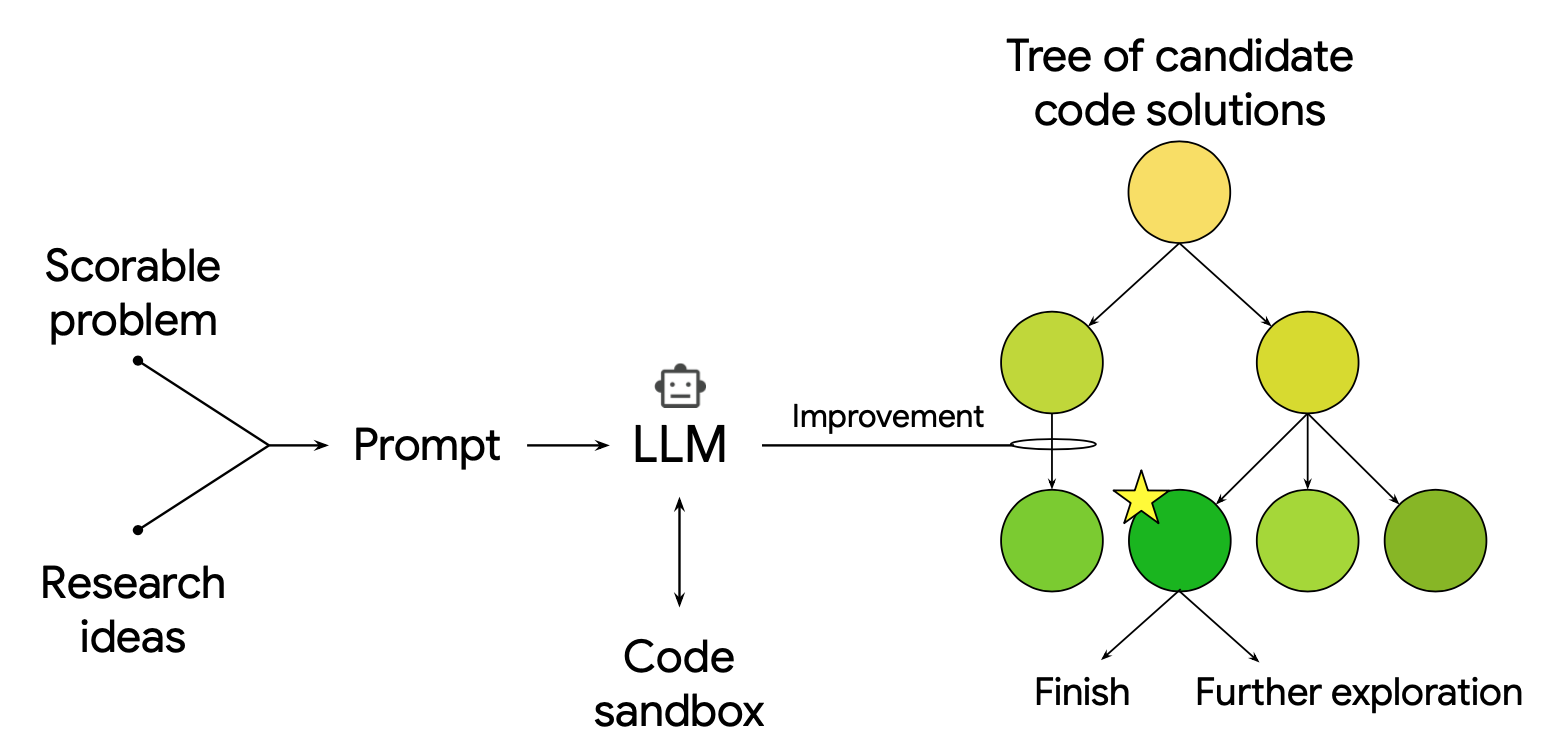
這個 AI 系統的核心是結合了大型語言模型和樹狀搜尋的策略,你可以將整個過程想像成一個「智慧程式碼生成器」。 首先,科學問題被轉化為一個「可評分任務」(scorable task),即能夠透過分數來衡量軟體表現的任務。 LLM 隨後會根據任務描述、先前的嘗試結果,以及從外部來源 1(例如頂尖論文或教科書)獲得的複雜研究想法,自動重寫並創建新的程式碼候選方案。
樹狀搜尋則扮演了總策略師的角色。 它不斷地評估這些候選方案的「品質分數」,並決定哪些方案值得進一步「探索」(嘗試新穎但風險高的方法)或「利用」(優化現有的好方法)。 這種迭代優化循環能夠不知疲倦地執行大規模的解決方案搜尋,找到那些像是「大海撈針」般的高品質解決方案。 這種方法類似於結合了強化學習概念(如 AlphaZero 2 3)的程式碼生成,但這裡的目標是優化科學任務的品質分數。
AI 在多領域的傑出表現
一、生物資訊學領域:單細胞 RNA 定序數據整合
在單細胞 RNA 定序 (scRNA-seq) 數據分析中,主要挑戰是移除跨樣本的實驗差異,同時保留足夠的生物學訊號(如細胞類型)5。 AI 系統在 Open Problems benchmark 6 上展示了卓越性能。 該任務的評估標準基於 13 個指標,這些指標量化了差異移除程度以及生物學變異的保留情況。 透過系統性的探索,AI 總共產生了 40種 不同的方法,這些方法在排行榜上的表現超越了所有現有已發表的解決方案。 性能的關鍵提升來自於「想法的重組」。
例如,表現最佳的 Batch Balanced K-Nearest Neighbors (BBKNN) 7 實作是將 BBKNN 與 ComBat 8 結合。 具體來說,它是在經 ComBat 校正後的 PCA embedding 上計算鄰近點,從而移除了全域性的線性批次相關變異。 這種協同混合 (synergistic hybridization) 的能力,證明了 AI 能夠有效地整合現有方法的優勢,以實現超越單一方法的性能。
二、公共衛生領域:預測 COVID-19 住院人數
在公共衛生領域,AI 系統挑戰了 CovidHub 9 benchmark,目標是預測美國的 COVID-19 住院人數。 在一項涵蓋 2024 年至 2025 年的回溯性研究中,AI 生成的「Google Retrospective」模型,其平均加權區間分數 (weighted interval score, WIS) 為 26,表現優於官方標準模型 CovidHub Ensemble 的 29 分。
AI 系統的探索共產生了 14種 超越官方模型的策略,此舉主要來自於模型架構的混合與概念創新。 例如,將基於疾病傳播理論的流行病學模型 CEPH-Rtrend_covid 與統計學自迴歸模型 UMass-ar6_pooled 結合,產生了一個既有理論基礎又能對最新數據趨勢高度反應的混合模型。 此外,系統還能透過 Deep Research 10 和 AI co-scientist 11 生成全新的概念性策略,例如引入「反事實模擬」 (Counterfactual Simulation),透過運行數千次蒙地卡羅模擬(Monte Carlo simulations)來量化不確定性。
三、地理空間分析:遙感影像語義分割
地理空間分析領域的挑戰是遙感影像的語義分割,這項電腦視覺任務要求對影像中的每個像素賦予特定的類別標籤。 Dense Labeling Remote Sensing Dataset (DLRSD) 12 是一個涉及 17 個類別標籤的密集標註數據集。 由於時間、季節、天氣和物體本身(例如建築物)的高度多樣性,該任務面臨著巨大的視覺異質性難度。
AI 系統產生的三個頂級解決方案顯著優於近期學術論文報告的結果,達到了平均交集聯合度 (mIoU) 超過 0.80 的成績。 這些成功的方案在架構上採用了 UNet++ 或 SegFormer(一種基於 Transformer 的架構),並結合了強大的編碼器(如 efficientnet-b7 或 se-resnext101-32x4d)。 另一個關鍵的策略是廣泛應用了 測試時增強 (Test-Time Augmentation, TTA),透過預測單個測試影像的多個增強版本(如水平翻轉、旋轉)的遮罩並將其平均,從而提高了預測的穩健性。
四、神經科學:腦神經活動預測
斑馬魚大腦神經活動預測 ZAPBench 13 旨在測試整個大腦(超過 7 萬個神經元)的神經活動。 AI 系統根據過去 4 個 time steps 的神經元活動來預測所有神經元未來 32 個 time steps 的活動。 結果顯示,AI 生成的模型表現優於所有現有的時間序列和影片預測方法,甚至超越了表現最佳的影片模型。 除了優異的準確性外,AI 解決方案的訓練速度也快了幾個數量級。 例如,AI 的時間序列解決方案在單個 T4 GPU 上訓練時間不到兩小時,而表現最佳的影片模型則需要在 16 個 A100 GPU 上訓練 36 小時。
五、時間序列預測:GIFT-Eval
General Time Series Forecasting Model Evaluation (GIFT-Eval) 14 基準測試涵蓋了來自七個不同領域的 28 個數據集,時間頻率從秒到年不等。 AI 系統進行兩種實驗:一種是針對每個數據集的獨立解決方案,另一種是創建一個通用的預測模型。
最終的預測模型結合了一個通用的 library 與八種配置。 透過在驗證集上挑選最佳配置並應用於測試集,模型能在不接觸測試資料的情況下自動調整策略。 該模型經多次迭代改進,從初始 MASE 0.82 的基準模型出發,最終經 500 個節點的精煉後達到 MASE 0.734。 其核心作法是依序建模並移除時序中的基礎部分,並將各部分的預測結果相加,從而系統地處理資料中的多種變異來源。
六、數值分析:解決困難積分
在數值分析領域,AI 的任務是解決標準演算法難以處理的困難積分,特別是那些具有無窮上限的振盪積分 (oscillatory integral)。 該任務的目標是找到一種通用方法,其性能優於廣泛使用的標準函式庫 scipy.integrate.quad() 15。 研究團隊挑選 38 個套用在 quad() 函式庫上失敗的積分,一半用於訓練、一半用於評估。
AI 生成的程式碼成功地將作為評估的 19 個積分中的 17 個計算至 3% 誤差內。 該解決方案的創新在於它結合了標準的 quad() 函式庫與級數加速技術。 它首先將無窮積分域劃分為一系列有限的子區間,從而將定積分轉化為一個無窮級數。 隨後,它應用了強大的級數加速技術,即尤拉轉換 (Euler's transformation),透過對初始項的重複平均來推斷緩慢收斂級數的極限,從而獲得積分真值的精確估計。 AI 的程式碼會優先使用 quad(),只有當 quad() 返回高誤差估計或失敗時,才會退而求其次使用其更專業的方法。
結語
這種結合 LLM 與樹狀搜尋的 AI 系統相當有潛力。 對於科學家而言,它將實驗探索週期從數週或數月縮短到數小時或數天。 這意味著研究人員可以更快地測試複雜假設、篩選數十種不同的方法,並將更多精力投入到結果分析和新概念發想上。 任何可以量化為「品質分數」的科學問題——從藥物標靶發現到氣候建模——都可能因這個系統而迎來革命性的加速。
儘管這個 AI 系統展示了傑出的成果,我們仍需誠實面對其侷限性。 首先,它僅限於解決那些可以被明確定義的可評分任務,對於缺乏清晰量化指標或需要大量開放式探索的科學問題則難以適用。 其次,如同傳統的遺傳編程 (Genetic Programming, GP) 16 和 AutoML 17,系統的有效性依賴於有效的搜索策略和 LLM 整合外部知識的能力。
然而,Google 開發的這個 AI 系統依然在科學軟體領域中畫下了一個里程碑。 它證明了 AI 能夠系統性地創新、組合和優化專家級的解決方案,並在數十個關鍵的科學難題上超越了人類專家的成果,大幅縮短了科學探索的耗時。 研究人員相信,至少在可評分領域,科學進步正處於革命性加速的前夕。 未來,AI 將不再只是科學家的工具,更可能成為一位不知疲倦、富有創意的協作者,極大地推動人類知識的疆界。
Footnotes
B. Romera-Paredes et al., “Mathematical discoveries from program search with large language models,” Nature, vol. 625, pp. 468–475, 2024. ↩
D. Silver et al., “Mastering the game of Go with deep neural networks and tree search,” Nature, vol. 529, pp. 484–489, 2016. ↩
D. Silver et al., “Mastering the game of Go without human knowledge,” Nature, vol. 550, pp. 354–359, 2017. ↩
E. Aygün et al., “An AI system to help scientists write expert-level empirical software,” Sep. 08, 2025, arXiv:2509.06503. ↩
Stuart, T. & Satija, R. Integrative single-cell analysis. Nat. Rev. Genet. 20, 257–272 (2019). ↩
Luecken, M. D. et al. Defining and benchmarking open problems in single-cell analysis. Nat. Biotechnol. 43, 1035–1040 (2025). ↩
K. Polański et al., “BBKNN: fast batch alignment of single cell transcriptomes,” Bioinformatics, vol. 36, pp. 964–965, 2019. ↩
W. E. Johnson, C. Li, & A. Rabinovic, “Adjusting batch effects in microarray expression data using empirical Bayes methods,” Biostatistics, vol. 8, pp. 118–127, 2007. ↩
Centers for Disease Control and Prevention. COVID-19 forecast hub (2025). URL https://github.com/cdcgov/covid19-forecast-hub?tab=readme-ov-file. ↩
Google. Gemini Deep Research (2025). URL https://gemini.google/overview/deep-research/?hl=en. ↩
J. Gottweis et al., “Towards an AI co-scientist,” arXiv preprint arXiv:2502.18864, 2025. ↩
Z. Shao, K. Yang, & W. Zhou, “Performance evaluation of single-label and multi-label remote sensing image retrieval using a dense labeling dataset,” Remote Sens., vol. 10, p. 964, 2018. ↩
J.-M. Lueckmann et al., “ZAPBench: a benchmark for whole-brain activity prediction in zebrafish,” arXiv preprint arXiv:2503.02618, 2025. ↩
T. Aksu et al., “GIFT-Eval: a benchmark for general time series forecasting model evaluation,” arXiv preprint arXiv:2410.10393, 2024. ↩
R. Piessens et al., QUADPACK: a subroutine package for automatic integration. Springer-Verlag, 1983. ↩
J. R. Koza, “Genetic programming as a means for programming computers by natural selection,” Stat. Comput., vol. 4, pp. 87–112, 1994. ↩
F. Hutter, L. Kotthoff, & J. Vanschoren, Automated machine learning: methods, systems, challenges. Springer Nature, 2019. ↩