- Published on
CLIP & CLASH 定序實驗技術
- Authors

- Name
- Ryan Chung
CLIP
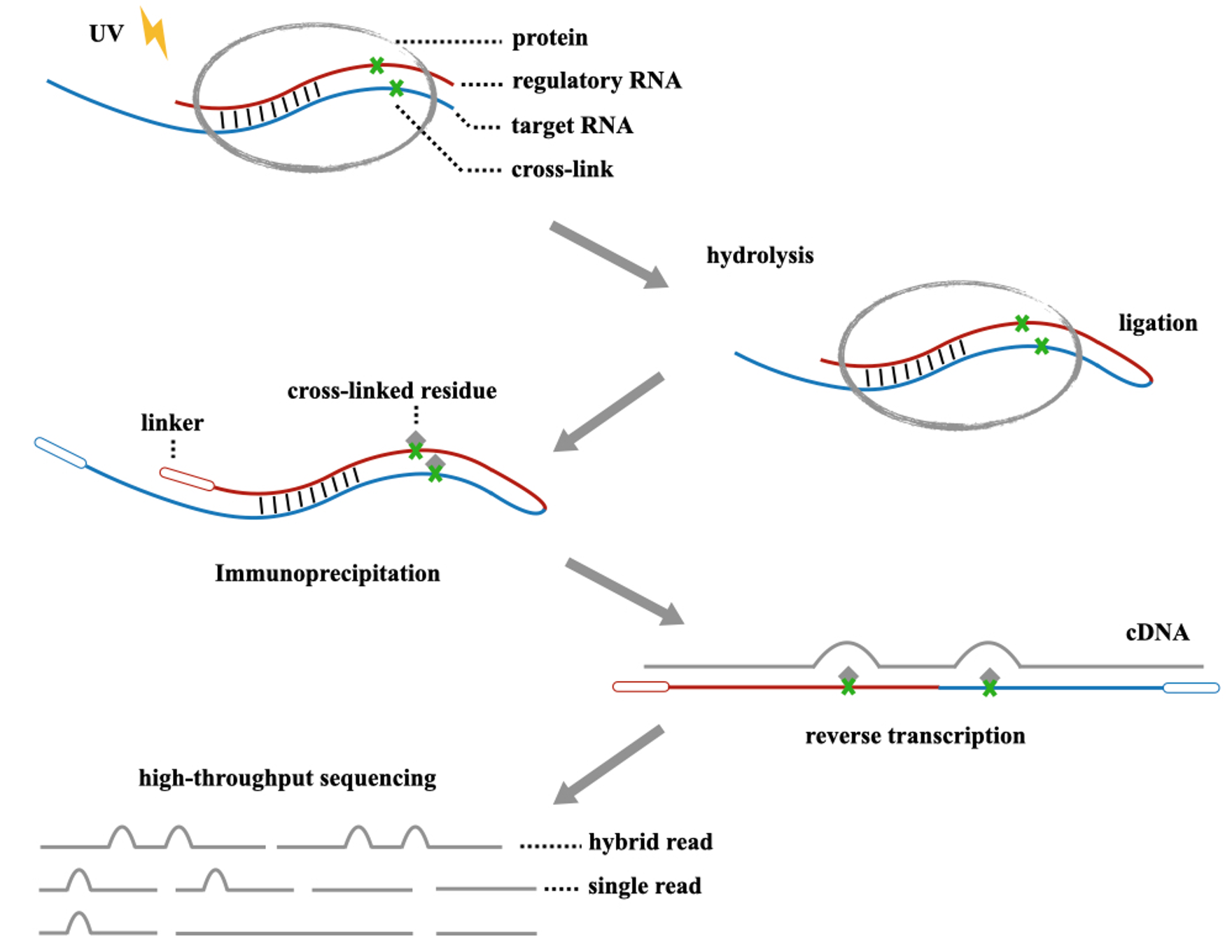
CLIP (UV-crosslinking and immunoprecipitation) 是研究 蛋白質與 RNA 交互作用 的重要技術,於 2003 年被 Darnell 等人發明。 CLIP 的原理主要是利用蛋白質與 RNA 會在 256nm 紫外光照射下發生共價交聯之特性,之後 CLIP 又衍生出許多不同的技術,例如: CRAC、PAR-CLIP (photoactivatable-ribonucleoside-enhanced CLIP,提高交聯效率)、CLIP-seq (crosslinking-immunprecipitation and high-throughput sequencing,CLIP 結合高通量定序技術)等,但大致上基本流程皆相同。1
CLIP 基本技術流程如圖上半部所示、CLIP-Seq 流程則如完整圖示。
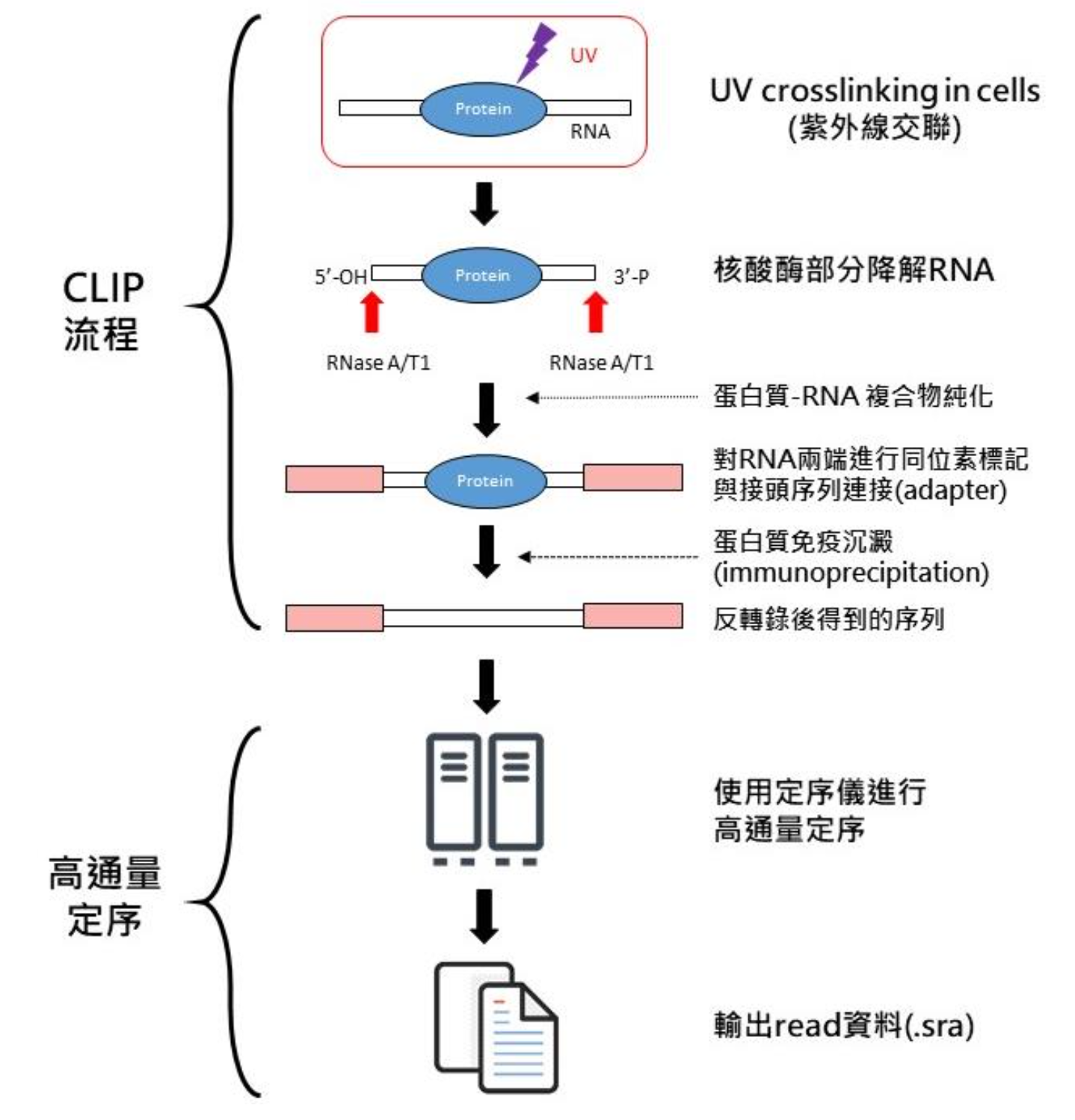
CLIP-Seq 資料的分析通常需要實驗組與對照組相互比對,實驗組的內容是將與蛋白質-RNA 結合對抓下來後使用免疫沉澱將蛋白質去除,將剩下的會與蛋白質結合之 RNA 去做定序並給定序列出現過的次數,稱為 read count;對照組的內容為直接打破細胞核中後對其中的 RNA 片段做定序並給定 read count。
名詞解釋
- Cross-link 鏈結
In chemistry and biology a cross-link is a bond or a short sequence of bonds that links one polymer chain to another. These links may take the form of covalent bonds or ionic bonds and the polymers can be either synthetic polymers or natural polymers (such as proteins).
簡單來說就是聚合物的共價鍵連結,crosslinking 就是製造鏈結,此處使用 UV 紫外光照射來製造聚合物
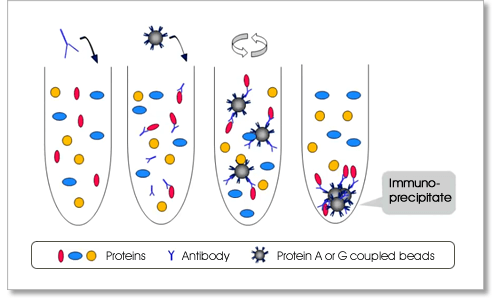
- Immunoprecipitation, IP 免疫沉澱法
一種將蛋白質視為抗原,並利用抗體與之進行特異性結合的特性,來進行研究。這項技術可用來將含有上千種不同蛋白質的樣品中,分離和濃縮出特定蛋白質。
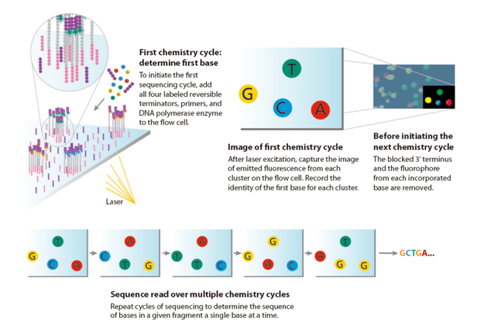
- 次世代基因定序(Next Generation Sequencing, NGS)
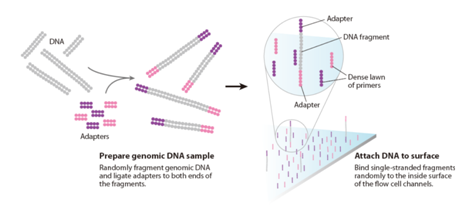
傳統 Sanger 方法進行基因組定序時,必須先將目標基因一段一段的增幅放大,一段讀長約 1000bp,定序完後再進行比對與組裝,操作繁瑣且費時費工費錢;NGS 以基因工程的技術將基因組核酸切成小片段,並接上轉接子 (adapter),藉由材料科學與影像系統的協助來進行快速且高通量的定序。不同技術平台之片段讀長略為不同,通常在 400bp 以下,後續藉由功能強大的資訊軟硬體來進行序列的片段接合,以及基因組的比對與組裝。
Illumina TruSeq 舉例:
- 將待測 DNA 打斷成 300-500bp 片段,並於兩端接上 adapter。
- 將已接上 adapter 的片段,loding 到表面帶有互補 adapter 序列的晶片 (flow cell) 上。
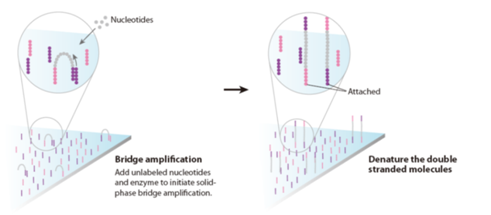
- 透過橋式聚合酶鏈鎖反應進行增幅。
- loding 不同鹼基且標記特定可移除螢光分子的去氧核糖核苷酸 (dNTP) 與反應試劑,重覆進行螢光標記移除與偵測,以達到快速且大量的定序結果。
CLASH
CLASH (Cross-linking ligation and sequencing of hybrids) 實驗技術是由 CLIP- Seq 改良而來的,如圖所示,用於高通量定序實驗中鑑定 RNA-RNA 的相互作用,和 CLIP-Seq 最大的不同在於 CLASH 可以更進一步的抓出與 Protein-RNA 結合的 RNA 片段,RNA-RNA 相互作用在基因調控、RNA 修飾等過程中發揮很大的作用。2
近年來 CLASH 技術在 miRNA 與 mRNA 的交互作用的實驗上有重大的貢獻,miRNA 與 mRNA 的交互作用發生在與 AGO 蛋白結合的狀態下,利用 CLASH 技術可以成功抓出會產生交互作用的 miRNA-mRNA 對。3
步驟
- 利用紫外光照射,使 RNA 與蛋白質之間產生共價交聯(Crosslink),使其複 合物結合更加緊密。
- 利用核酸酶降解未被蛋白質結合而裸露的 RNA 序列(非真實結合位點以外序列),並以酵素將兩作用 RNA 片段相黏 (Ligation)。
- 利用抗體將 protein-RNA-RNA 複合物進行蛋白質免疫沉澱純化 (Immunoprecipitation),以萃取出帶有兩者資訊之序列 (hybrid RNA),並在兩端黏上 Linker (adapter)
- 最後,將序列反轉錄複製成 cDNA 後進行高通量定序,即可得到帶有相互作用的 RNA 序列片段 (CLASH read)。
名詞解釋
- CLASH
CLASH maps RNA-RNA interactions. In this method, RNA-protein complexes are UV-crosslinked and affinity-purified. RNA-RNA hybrids are ligated, isolated, and reverse-transcribed into cDNA. Deep sequencing of the cDNA provides high-resolution chimeric reads of RNA-RNA interactions.

Ligation 連接
In molecular biology, ligation is the joining of two nucleic acid fragments through the action of an enzyme. It is an essential laboratory procedure in the molecular cloning of DNA whereby DNA fragments are joined together to create recombinant DNA molecules, such as when a foreign DNA fragment is inserted into a plasmid.
通過酶的作用連接 DNA 或 RNA 片段
cDNA
Complementary DNA ( 互補DNA ),是以 RNA 為模板反轉錄成的複製品
兩者比較
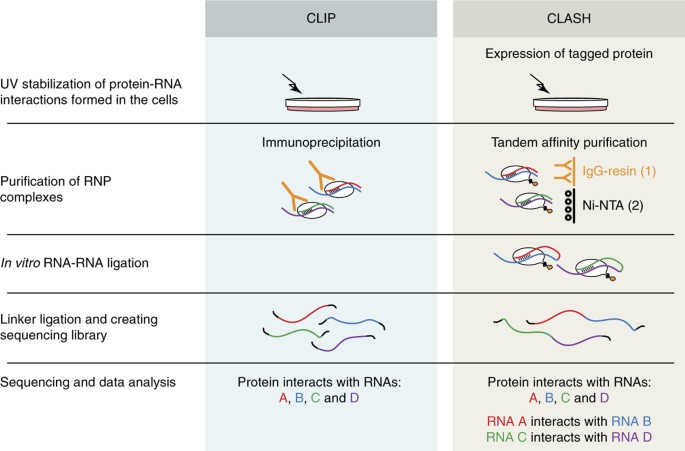
圖片來源為 CLASH 原始論文 2
補充
- CLASH 製備過程可能在 ligation 時沒有接合好,造成結果中的 reads 不一定只有 RNA-RNA pair,也可能存在斷裂的單獨 RNA
- CLASH 實驗過程可能將兩個不相關序列 ligate 上(非真實結合事件)
- CLASH 實驗包含大量 non-canonical binding,大部分不具備基因調控功能
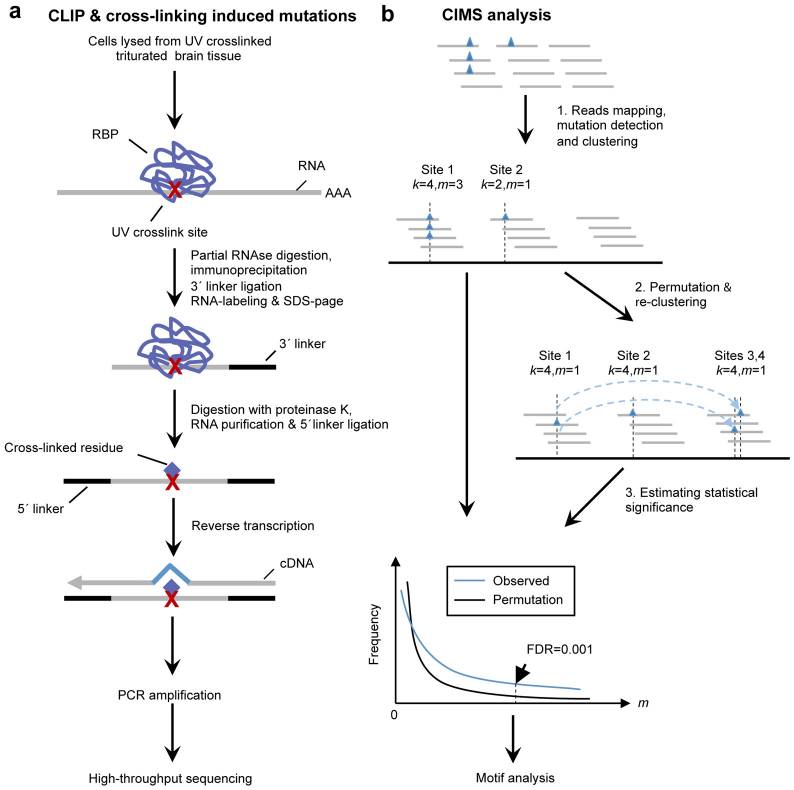
- CLIP 或 CLASH 實驗過程可能因為蛋白質降解不完全,機率性 (8–20%) 造成 crosslink 處發生突變 Crosslinking Induced Mutation Sites (CIMS),並經由 PCR 放大出現在大量的序列中 4
Footnotes
M. Hafner, M. Katsantoni, T. Köster, J. Marks, J. Mukherjee, and D. Staiger, “CLIP and complementary methods,” Nature Reviews Methods Primers, vol. 1, no. 1, Mar. 2021, doi: https://doi.org/10.1038/s43586-021-00018-1. ↩
A. Helwak and D. Tollervey, “Mapping the miRNA interactome by cross-linking ligation and sequencing of hybrids (CLASH),” Nature Protocols, vol. 9, no. 3, pp. 711–728, Feb. 2014, doi: https://doi.org/10.1038/nprot.2014.043. ↩ ↩2
A. Helwak, G. Kudla, T. Dudnakova, and D. Tollervey, “Mapping the Human miRNA Interactome by CLASH Reveals Frequent Noncanonical Binding,” Cell, vol. 153, no. 3, pp. 654–665, Apr. 2013, doi: https://doi.org/10.1016/j.cell.2013.03.043. ↩
Zhang C, Darnell RB, "Mapping in vivo protein-RNA interactions at single-nucleotide resolution from HITS-CLIP data," Nature biotechnology, vol. 29, no.7, pp. 607-614, Jun 2011, doi:10.1038/nbt.1873. ↩